Nikotinamida adenina dinukleotida

| |
| Nama | |
|---|---|
| Nama lain
Difosfopiridina nukleotida (DPN+), Koenzim I
| |
| Penanda | |
Model 3D (JSmol)
|
|
| 3DMet | {{{3DMet}}} |
| ChEBI | |
| ChemSpider | |
| Nomor EC | |
| KEGG | |
PubChem CID
|
|
| Nomor RTECS | {{{value}}} |
| |
| Sifat | |
| C21H27N7O14P2 | |
| Massa molar | 663,43 g/mol |
| Penampilan | bubuk putih |
| Titik lebur | 160 °C |
| Bahaya | |
| Bahaya utama | Tidak berbahaya |
Kecuali dinyatakan lain, data di atas berlaku pada suhu dan tekanan standar (25 °C [77 °F], 100 kPa). | |
| Referensi | |
Nikotinamida adenina dinukleotida, disingkat NAD+, adalah koenzim yang ditemukan di semua sel hidup. Senyawa ini berupa dinukleotida, yakni mengandung dua nukleotida yang dihubungkan melalui gugus fosfat, dengan satu nukleotida mengandung basa adenina dan yang lainnya mengandung nikotinamida.
Dalam metabolisme, NAD+ terlibat dalam reaksi redoks, dengan membawa elektron dari satu reaksi ke reaksi lainnya. Koenzim ini oleh karenanya ditemukan dalam dua bentuk yang berbeda: NAD+ sebagai oksidator, dan NADH sebagai reduktor. NAD+ menerima elektron dari molekul lain dan menjadi tereduksi (NADH), dan begitu pula sebaliknya. Reaksi transfer elektron ini merupakan salah satu fungsi NAD+. Namun ia juga memiliki fungsi lain pada proses selular lainnya, utamanya adalah sebagai substrat enzim yang menambah maupun mengurangi gugus fungsi pada protein dalam modifikasi pascatranslasional. Karena fungsinya yang penting ini, enzim-enzim yang terlibat dalam metabolisme sering menjadi target pengembangan obat-obatan.
Dalam organisme, NAD+ dapat disintesis secara de novo (dari blok-blok molekul kecil) dari asam amino triptofan ataupun asam aspartat. Selain itu, NAD+ dapat juga diperoleh dari sumber makanan yang mengandung vitamin niasin.
Sifat-sifat fisika dan kimia
Nikotinamida adeninan dinukleotida, sama seperti senyawa dinukleotida pada umumnya, mengandung nukleotida yang dihubungkan oleh satu pasang gugus fosfat yang menjembatani keduanya. Nukleotida ini tersusun atas cincin ribosa dengan adenina yang melekat pada atom karbon pertama 1' cincin tersebut. Gugus nikotinamida dapat dilekatkan ke dalam dua orientasi pada satu atom karbon anomerik. Karena terdapat dua struktur yang dimungkinkan, senyawa ini berupa diastereomer. Diastereomer β-nikotinamida dari NAD+ adalah bentuk yang ditemukan pada organisme. Kedua nukleotida ini dihubungkan bersama oleh dua gugus fosfat melalui karbon 5'.[1]

Dalam metabolisme, senyawa ini menerima ataupun mendonorkan elektronnya dalam reaksi redoks.[2] Reaksi ini (diringkaskan oleh persamaan di bawah) melibatkan pelepasan dua atom hidrogen dari reaktan (R), dalam bentuk ion hidrida (H−) dan proton (H+). Proton dilepaskan ke dalam larutan, manakala reduktan RH2 dioksidasi dan NAD+ direduksi menjadi NADH melalui transfer hidrida menuju cincin nikotinamida.
- RH2 + NAD+ → NADH + H+ + R
Dari pasangan elektron hidrida, satu elektron ditransfer ke nitrogen cincin nikotinamida yang bermuatan positif, dan atom hidrogen kedua di transfer ke atom karbn C4 yang berseberangan dengan nitrogen ini. Potensial titik tengah pasangan redoks NAD+/NADH adalah −0,32 volt, membuat NADH sebagai reduktor kuat.[3] Reaksi ini sangat mudah berbalik arah, ketika NADH direduksi menjadi molekul lain dan dioksidasi kembali menjadi NAD+. Hal ini berarti koenzim ini dapat secara terus menerus berada dalam siklus NAD+ dan NADH tanpa sendirinya dikonsumsi.[1]
Secara fisik, koenzim ini berbentuk bubuk amorf yang higroskopik dan sangat larut dalam air.[4] Padatan ini stabil jika disimpan dalam keadaan gelap dan kering. Larutan NAD+ tidak berwarna dan stabil selama satu pada temperatur 4 °C dan pH netral. Ia akan terurai dengan cepat apabila terkena asam ataupun basa. Seketika terurai, produk dekomposisi ini merupakan inhibitor enzim.[5]
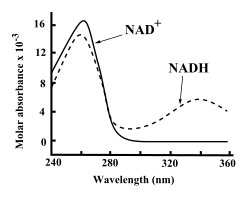
Baik NAD+ dan NADH menyerap ultraviolet dengan sangat kuat oleh karena keberadaan basa adeninanya. Sebagai contoh, puncak absorpsi NAD+ berada pada panjang gelombang 259 nanometer (nm), dengan koefisien pemunahan 16.900 M−1cm−1. NADH juga menyerap panjanga gelombang yang lebih tinggi, dengan puncak kedua dalam absorpsi UV-nya adalah 339 nm dengan koefisien pemunahan 6.220 M−1cm−1.[6] Perbedaan spektrum absorpsi ultraviolet antara bentuk koenzim yang teroksidasi dengan yang tereduksi ini membuat zat ini sangat mudah diukur pada asai enzim.[6]
NAD+ dan NADH juga memiliki spektrum fluoresens yang berbeda. NADH dalam larutan memiliki puncak emisi pada 460 nm dan paruh waktu fluoresens sepanjang 0,4 nanosekon, manakala pada koenzim yang teroksidasi tidak memiliki emisi fluoresens.[7] Ciri-ciri sinyal fluoresens berubah ketika NADH mengikat kepada protein, sehingga perubahan ini dapat digunakan untuk mengukur tetapan disosiasi, yang sangat berguna dalam kajian kinetika enzim.[7][8] Perubahan dalam sinyal fluoresens ini juga digunakan untuk mengukur perubahan dalam keadaan redoks sel hidup, melalui mikroskopi fluoresens.[9]
Konsentrasi dan keadaan zat dalam sel
Dalam hati tikus, kandungan total NAD+ dan NADH adalah kira-kira 1 μmol per gram berat basah hewan, sekitar 10 kali lipat konsentrasi NADP+ dan NADPH dalam sel yang sama.[10] Konsentrasi sebenarnya NAD+ dalam sitosol sel sulit diukur, dengan perkiraan terbaru dalam sel hewan berkisar antara 0,3 M[11][12] sampai dengan 1,0 sampai 2,0 mM dalam ragi.[2] Namun, sekitar 80% zat ini terikat pada protein, sehingga konsentrasinya dalam larutan haruslah lebih rendah.[13]
Data konsentrasi zat ini pada bagian sel lainnya sangat terbatas, walaupun dalam mitokondria konsentrasi NAD+ sama dengan konsentrasi zat ini dalam sitosol.[12] NAD+ dibawa ke dalam mitokondria melalui protein transpor membran yang khusus oleh karena koenzim ini tidak dapat berdifusi melewati membran.[14]
Keseimbangan antara bentuk yang teroksidasi dengan bentuk yang tereduksi disebut sebagai rasio NAD+/NADH. Rasio ini adalah komponen penting yang disebut sebagai keadaan redoks sel. Keadaan redoks sel adalah pengukuran yang mencerminkan baik aktivitas metabolisme sel maupun kesehatan sel.[15] Efek rasio NAD+/NADH terhadap sel sangatlah kompleks. Ia mengontrol aktivitas beberapa enzim kunci, meliputi gliseraldehida 3-fosfat dehidrogenase dan piruvat dehidrogenase. Dalam jaringan sel mamalia yang sehat, perkiraan rasio NAD+/NADH umumnya berkisar sekitar 700; rasio ini oleh karenanya sangat memfavoritkan reaksi oksidasi.[16][17] Sebaliknya, rasio NADP+/NADPH umumnya sekitar 0,005.[18]
Biosintesis
NAD+ disintesis melalui dua lintasan metabolisme. Ia diproduksi baik melalui lintasan de novo yang menggunakan asam amino maupun melalui lintasan daur ulang dengan mendaur ulang komponen-komponen prekursor seperti nikotinamida menjadi NAD+, lintasan yang lain disebut lintasan kinurenina yang dilalui oleh TRP.[19] Lintasan kinurenina terbagi dua, yang pertama adalah lintasan asam kinurenat, yang kedua adalah lintasan asam kuinolinat dan hidroksikynurenina-3. Ketiga senyawa organik tersebut merupakan prekursor dari NAD+.
Produksi De novo

Kebanyakan organisme mensintesis NAD+ dari komponen-komponen yang sederhana.[2] Reaksi yang terlibat berbeda-beda dari organisme yang satu ke organisme lain. Namun terdapat kesamaan dalam penghasilan QA dari asam amino tertentu seperti TRP pada hewan dan beberapa bakteri, ataupun asam aspartat pada beberapa bakteri dan tumbuhan.[20][21] Asam kuinolinat diubah menjadi asam nikotinat adenina dinukleotida (NaMN) melalui transfer gugus fosforibosa. Gugus adenilat kemudian ditransfer untuk membentuk asam nikotinat adenina dinukleotida (NaAD). Pada akhirnya, gugus asam nikotinat pada NaAD diamidasi menjadi gugus nikotinamida (Nam), membentuk nikotinamida adenina dinukleotida.[2]
Pada langkah yang lebih lanjut, beberapa NAD+ diubah menjadi NADP+ oleh NAD+ kinase, yang memfosforilasi NAD+.[22] Pada kebanyakan organisme, enzim ini menggunakan ATP sebagai sumber gugus fosfat, walaupun pada bakteri seperti Mycobacterium tuberculosis dan archaea seperti Pyrococcus horikoshii menggunakan polifosfat anorganik sebagai donor fosfat alternatif.[23][24]

Lintasan daur ulang
Selain perakitan NAD+ secara de novo menggunakan asam amino sederhana, sel juga mendaur ulang senyawa-senyawa yang mengandung nikotinamida untuk menghasilkan NAD+. Walaupun terdapat banyak prekursor-prekursor yang diketahui, terdapat tiga senyawa alamiah mengandung cincin nikotinamida yang digunakan dalam lintasan daur ulang ini, yakni asam nikotinat (Na), nikotinamida (Nam), dan nikotinamida ribosida (NR).[25] Prekursor-prekursor ini kemudian dimasukkan ke dalam lintasan biosintesis NAD(P)+ melalui reaksi adenilasi dan fosforibosilasi seperti yang ditunjukkan pada ilustrasi di atas.[2] Senyawaan-senyawaan ini dapat berasal dari makanan, di mana campuran asam nikotinat dan nikotinamida disebut sebagai vitamin B3 ataupun niasin. Namun, senyawa-senyawa ini juga dapat diproduksi dalam sel sendiri, yaitu melalui pelepasan gugus nikotinamida dari NAD+ dalam reaksi transfer ADP-ribosa. Enzim-enzim yang terlibat dalam lintasan daur ulang ini tampaknya terkonsentrasi dalam inti sel, yang mengompensasikan laju konsumsi NAD+ yang tinggi dalam organel ini.[26] Sel juga dapat mendapatkan NAD+ secara ekstraseluler (luar sel) dari sekelilingnya.[27]
Walaupun terdapat lintasan de novo, lintasan daur ulang ini merupakan lintasan yang esensial pada manusia. Kekurangan niasin pada makanan mengakibatkan penyakit defisiensi vitamin pelagra.[28] Kebutuhan NAD+ yang tinggi ini disebabkan oleh konsumsinya yang tinggi pada reaksi modifikasi pascatranslasi.[2]
Lintasan daur ulang yang digunakan oleh mikroorganisme berbeda dengan lintasan yang digunakan oleh mamalia.[29] Beberapa patogen seperti ragi Candida glabrata dan bakteri Haemophilus influenzae adalah auksotrof NAD+ (yakni tidak dapat mensintesis NAD+). Namun mereka memiliki lintasan daur ulang, sehingga sangat bergantung pada sumber luar NAD+ dan prekursornya.[30][31] Bahkan pada patogen seperti Chlamydia trachomatis, ia tidak memiliki gen untuk biosintesis maupun daur ulang NAD+ dan NADP+, sehingga harus menerima asupan koenzim ini dari sel inangnya.[32]
Fungsi

Nikotinamida adenina dinukleotida memiliki beberapa peranan esensial dalam metabolisme. Ia berperan sebagai koenzim pada reaksi redoks, sebagai donor gugus ADP-ribosa pada reaksi ADP-ribosilasi, sebagai prekursor molekul penghantara kedua ADP-ribosa siklik, dan juga sebagai substrat bagi enzim DNA ligase bakteri dan enzim sirtuin yang menggunakan NAD+ untuk melepaskan gugus asetil dari protein.
Oksidoreduktase
Peran utama NAD+ dalam metabolisme adalah mentransfer elektron dari satu molekul ke molekul lainnya. Reaksi seperti ini dikatalisasi oleh sekelompok besar enzim yang dinamakan oksidoreduktase. Tata nama enzim dalam kelompok oksidoreduktase mengandung nama kedua substratnya. Sebagai contoh, NADH-ubikuinon oksidoreduktase mengkatalisis oksidasi NADH oleh koenzim Q.[34] Namun, enzim oksidoreduktase ini juga dapat dirujuk sebagai dehidrogenase ataupun reduktase. Biasanya NADH-ubikuinon oksidoreduktase disebut sebagai NADH dehidrogenase ataupun kadang kala koenzim Q reduktase.[35]
Ketika terikat pada suatu protein, NAD+ dan NADH biasanya terikat pada motif struktural yang dikenal dengan nama lipatan Rossmann.[36] Motif ini dinamakan atas nama Michael Rossmann yang merupakan ilmuwan yang pertama kali memperhatikan banyaknya motif ini pada protein pengikat nukleotida.[37] Lipatan ini mengandung tiga atau lebih lempengan beta paralel yang dihubungkan oleh dua heliks alfa dengan urutan beta-alfa-beta-alfa-beta. Oleh karena tiap lipatan Rossmann mengikat satu nukleotida, domain pengikatan untuk dinukleotida NAD+ terdiri dari dua lipatan Rossmann yang berpasangan, dengan tiap lipatan mengikat satu nukleotida.[37] Walau demikian, lipatan ini tidaklah universal ada pada enzim yang bergantung pada NAD. Baru-baru ini ditemukan suatu kelas enzim bakteria yang terlibat dalam metabolisme asam amino mengikat koenzim ini, namun tidak memiliki motif lipatan Rossmann.[38]

Ketika terikat pada tapak aktif suatu oksidoreduktase, cincin nikotinamida koenzim ini diposisikan sedemikiannya ia dapat menerima hidrida dari substrat enzim lainnya. Oleh karena karbon C4 yang menerima hidrogen ini prokiral, hal ini dapat digunakan dalam kinetika enzim untuk mengetahui mekanisme enzim. Hal ini dilakukan dengan mencampurkan enzim dengan substrat yang beratom deuterium sebagai pengganti hidrogen, sehingga enzim akan mereduksi NAD+ dengan mentransfer deuterium daripada hidrogen. Dalam kasus ini, enzim dapat menghasilkan salah satu stereoisomer NADH. Pada beberapa jenis enzim, hidrogen ditransfer dari atas bidang cincin nikotinamida. Enzim demikian disebut sebagai oksidoreduktase kelas A, manakala enzim kelas B mentransfer atom hidrogennya dari bawah bidang.[39]
Walaupun terdapat kemiripan pada cara protein mengikat koenzim NAD+ dan NADP+, enzim hampir selalu memiliki spesifisitas yang tinggi untuk mengikat hanya salah satu dari NAD+ maupun NADP+.[40] Spesifisitas ini mencermikan peranan metabolik kedua koenzim yang berbeda dan merupakan akibat dari perbedaan residu asam amino yang berbeda pada kantong pengikat koenzim tersebut. Sebagai contohnya, pada tapak aktif enzim pengikat NADP, ikatan ion terbentuk antara rantai samping asam amino basa dengan gugus fosfat NADP+ yang asam. Sebaliknya, pada enzim yang mengikat NAD, muatan kantongnya terbalik, menjauhkan NADP+ untuk berikatan dengannya. Walau demikian, terdapat pengecualian terhadap kaidah ini. Enzim seperti aldosa reduktase, glukosa-6-fosfat dehidrogenase, dan metilenatetrahidrofolat reduktase dapat menggunakan kedua enzim tersebut pada beberapa spesies organisme.[41]
Referensi
- ^ a b Pollak, N (2007). "The power to reduce: pyridine nucleotides—small molecules with a multitude of functions". Biochem. J. 402 (2): 205–18. doi:10.1042/BJ20061638. PMC 1798440
 . PMID 17295611.
. PMID 17295611.
- ^ a b c d e f Belenky P (2007). "NAD+ metabolism in health and disease" (PDF). Trends Biochem. Sci. 32 (1): 12–9. doi:10.1016/j.tibs.2006.11.006. PMID 17161604. Diakses tanggal 2007-12-23. Kesalahan pengutipan: Tanda
<ref>tidak sah; nama "Belenky" didefinisikan berulang dengan isi berbeda - ^ Unden G (1997). "Alternative respiratory pathways of Escherichia coli: energetics and transcriptional regulation in response to electron acceptors". Biochim. Biophys. Acta. 1320 (3): 217–34. doi:10.1016/S0005-2728(97)00034-0. PMID 9230919.
- ^ Windholz, Martha (1983). The Merck Index: an encyclopedia of chemicals, drugs, and biologicals (edisi ke-10th). Rahway NJ, US: Merck. hlm. 909. ISBN 911910271 Periksa nilai: length
|isbn=(bantuan). - ^ Biellmann JF, Lapinte C, Haid E, Weimann G (1979). "Structure of lactate dehydrogenase inhibitor generated from coenzyme". Biochemistry. 18 (7): 1212–7. doi:10.1021/bi00574a015. PMID 218616.
- ^ a b Dawson, R. Ben (1985). Data for biochemical research (edisi ke-3rd). Oxford: Clarendon Press. hlm. 122. ISBN 0-19-855358-7.
- ^ a b Lakowicz JR, Szmacinski H, Nowaczyk K, Johnson ML (1992). "Fluorescence lifetime imaging of free and protein-bound NADH". Proc. Natl. Acad. Sci. U.S.A. 89 (4): 1271–5. doi:10.1073/pnas.89.4.1271. PMC 48431
 . PMID 1741380.
. PMID 1741380.
- ^ Jameson DM, Thomas V, Zhou DM (1989). "Time-resolved fluorescence studies on NADH bound to mitochondrial malate dehydrogenase". Biochim. Biophys. Acta. 994 (2): 187–90. PMID 2910350.
- ^ Kasimova MR, Grigiene J, Krab K; et al. (2006). "The free NADH concentration is kept constant in plant mitochondria under different metabolic conditions". Plant Cell. 18 (3): 688–98. doi:10.1105/tpc.105.039354. PMC 1383643
 . PMID 16461578.
. PMID 16461578.
- ^ Reiss PD, Zuurendonk PF, Veech RL (1984). "Measurement of tissue purine, pyrimidine, and other nucleotides by radial compression high-performance liquid chromatography". Anal. Biochem. 140 (1): 162–71. doi:10.1016/0003-2697(84)90148-9. PMID 6486402.
- ^ Yamada K, Hara N, Shibata T, Osago H, Tsuchiya M (2006). "The simultaneous measurement of nicotinamide adenine dinucleotide and related compounds by liquid chromatography/electrospray ionization tandem mass spectrometry". Anal. Biochem. 352 (2): 282–5. doi:10.1016/j.ab.2006.02.017. PMID 16574057.
- ^ a b Yang H, Yang T, Baur JA, Perez E, Matsui T, Carmona JJ, Lamming DW, Souza-Pinto NC, Bohr VA, Rosenzweig A, de Cabo R, Sauve AA, Sinclair DA. (2007). "Nutrient-Sensitive Mitochondrial NAD+ Levels Dictate Cell Survival". Cell. 130 (6): 1095–107. doi:10.1016/j.cell.2007.07.035. PMID 17889652.
- ^ Blinova K, Carroll S, Bose S; et al. (2005). "Distribution of mitochondrial NADH fluorescence lifetimes: steady-state kinetics of matrix NADH interactions". Biochemistry. 44 (7): 2585–94. doi:10.1021/bi0485124. PMID 15709771.
- ^ Todisco S, Agrimi G, Castegna A, Palmieri F (2006). "Identification of the mitochondrial NAD+ transporter in Saccharomyces cerevisiae". J. Biol. Chem. 281 (3): 1524–31. doi:10.1074/jbc.M510425200. PMID 16291748.
- ^ Schafer F, Buettner G (2001). "Redox environment of the cell as viewed through the redox state of the glutathione disulfide/glutathione couple". Free Radic Biol Med. 30 (11): 1191–212. doi:10.1016/S0891-5849(01)00480-4. PMID 11368918.
- ^ Williamson DH, Lund P, Krebs HA (1967). "The redox state of free nicotinamide-adenine dinucleotide in the cytoplasm and mitochondria of rat liver". Biochem. J. 103 (2): 514–27. PMC 1270436
 . PMID 4291787.
. PMID 4291787.
- ^ Zhang Q, Piston DW, Goodman RH (2002). "Regulation of corepressor function by nuclear NADH". Science. 295 (5561): 1895–7. doi:10.1126/science.1069300. PMID 11847309.
- ^ Veech RL, Eggleston LV, Krebs HA (1969). "The redox state of free nicotinamide-adenine dinucleotide phosphate in the cytoplasm of rat liver". Biochem. J. 115 (4): 609–19. PMC 1185185
 . PMID 4391039.
. PMID 4391039.
- ^ (Inggris)"Mitochondria, metabolic disturbances, oxidative stress and the kynurenine system, with focus on neurodegenerative disorders". Department of Neurology, University of Szeged; Sas K, Robotka H, Toldi J, Vécsei L. Diakses tanggal 2010-07-31.
- ^ Katoh A, Uenohara K, Akita M, Hashimoto T (2006). "Early steps in the biosynthesis of NAD in Arabidopsis start with aspartate and occur in the plastid". Plant Physiol. 141 (3): 851–7. doi:10.1104/pp.106.081091. PMC 1489895
 . PMID 16698895.
. PMID 16698895.
- ^ Foster JW, Moat AG (1 March 1980). "Nicotinamide adenine dinucleotide biosynthesis and pyridine nucleotide cycle metabolism in microbial systems". Microbiol. Rev. 44 (1): 83–105. PMC 373235
 . PMID 6997723.
. PMID 6997723.
- ^ Magni G, Orsomando G, Raffaelli N (2006). "Structural and functional properties of NAD kinase, a key enzyme in NADP biosynthesis". Mini reviews in medicinal chemistry. 6 (7): 739–46. doi:10.2174/138955706777698688. PMID 16842123.
- ^ Sakuraba H, Kawakami R, Ohshima T (2005). "First archaeal inorganic polyphosphate/ATP-dependent NAD kinase, from hyperthermophilic archaeon Pyrococcus horikoshii: cloning, expression, and characterization". Appl. Environ. Microbiol. 71 (8): 4352–8. doi:10.1128/AEM.71.8.4352-4358.2005. PMC 1183369
 . PMID 16085824.
. PMID 16085824.
- ^ Raffaelli N, Finaurini L, Mazzola F; et al. (2004). "Characterization of Mycobacterium tuberculosis NAD kinase: functional analysis of the full-length enzyme by site-directed mutagenesis". Biochemistry. 43 (23): 7610–7. doi:10.1021/bi049650w. PMID 15182203.
- ^ Tempel W, Rabeh WM, Bogan KL; et al. (2007). "Nicotinamide riboside kinase structures reveal new pathways to NAD+". PLoS Biol. 5 (10): e263. doi:10.1371/journal.pbio.0050263. PMC 1994991
 . PMID 17914902.
. PMID 17914902.
- ^ Anderson RM, Bitterman KJ, Wood JG; et al. (2002). "Manipulation of a nuclear NAD+ salvage pathway delays aging without altering steady-state NAD+ levels". J. Biol. Chem. 277 (21): 18881–90. doi:10.1074/jbc.M111773200. PMID 11884393.
- ^ Billington RA, Travelli C, Ercolano E; et al. (2008). "Characterization of NAD Uptake in Mammalian Cells". J. Biol. Chem. 283 (10): 6367–74. doi:10.1074/jbc.M706204200. PMID 18180302.
- ^ Henderson LM (1983). "Niacin". Annu. Rev. Nutr. 3: 289–307. doi:10.1146/annurev.nu.03.070183.001445. PMID 6357238.
- ^ Rongvaux A, Andris F, Van Gool F, Leo O (2003). "Reconstructing eukaryotic NAD metabolism". Bioessays. 25 (7): 683–90. doi:10.1002/bies.10297. PMID 12815723.
- ^ Ma B, Pan SJ, Zupancic ML, Cormack BP (2007). "Assimilation of NAD(+) precursors in Candida glabrata". Mol. Microbiol. 66 (1): 14–25. doi:10.1111/j.1365-2958.2007.05886.x. PMID 17725566.
- ^ Reidl J, Schlör S, Kraiss A, Schmidt-Brauns J, Kemmer G, Soleva E (2000). "NADP and NAD utilization in Haemophilus influenzae". Mol. Microbiol. 35 (6): 1573–81. doi:10.1046/j.1365-2958.2000.01829.x. PMID 10760156.
- ^ Gerdes SY, Scholle MD, D'Souza M; et al. (2002). "From genetic footprinting to antimicrobial drug targets: examples in cofactor biosynthetic pathways". J. Bacteriol. 184 (16): 4555–72. doi:10.1128/JB.184.16.4555-4572.2002. PMC 135229
 . PMID 12142426.
. PMID 12142426.
- ^ Senkovich O, Speed H, Grigorian A; et al. (2005). "Crystallization of three key glycolytic enzymes of the opportunistic pathogen Cryptosporidium parvum". Biochim. Biophys. Acta. 1750 (2): 166–72. doi:10.1016/j.bbapap.2005.04.009. PMID 15953771.
- ^ "Enzyme Nomenclature, Recommendations for enzyme names from the Nomenclature Committee of the International Union of Biochemistry and Molecular Biology". Diakses tanggal 2007-12-06.
- ^ "NiceZyme View of ENZYME: EC 1.6.5.3". Expasy. Diakses tanggal 2007-12-16.
- ^ Lesk AM (1995). "NAD-binding domains of dehydrogenases". Curr. Opin. Struct. Biol. 5 (6): 775–83. doi:10.1016/0959-440X(95)80010-7. PMID 8749365.
- ^ a b Rao S, Rossmann M (1973). "Comparison of super-secondary structures in proteins". J Mol Biol. 76 (2): 241–56. doi:10.1016/0022-2836(73)90388-4. PMID 4737475.
- ^ Goto M, Muramatsu H, Mihara H; et al. (2005). "Crystal structures of Delta1-piperideine-2-carboxylate/Delta1-pyrroline-2-carboxylate reductase belonging to a new family of NAD(P)H-dependent oxidoreductases: conformational change, substrate recognition, and stereochemistry of the reaction". J. Biol. Chem. 280 (49): 40875–84. doi:10.1074/jbc.M507399200. PMID 16192274.
- ^ Bellamacina CR (1 September 1996). "The nicotinamide dinucleotide binding motif: a comparison of nucleotide binding proteins". FASEB J. 10 (11): 1257–69. PMID 8836039.
- ^ Carugo O, Argos P (1997). "NADP-dependent enzymes. I: Conserved stereochemistry of cofactor binding". Proteins. 28 (1): 10–28. doi:10.1002/(SICI)1097-0134(199705)28:1<10::AID-PROT2>3.0.CO;2-N. PMID 9144787.
- ^ Vickers TJ, Orsomando G, de la Garza RD; et al. (2006). "Biochemical and genetic analysis of methylenetetrahydrofolate reductase in Leishmania metabolism and virulence". J. Biol. Chem. 281 (50): 38150–8. doi:10.1074/jbc.M608387200. PMID 17032644.
